AI预测酶的工作效率
酶在细胞代谢过程中发挥着关键作用。为了能够对这些过程进行定量评估,研究人员需要知道酶的所谓“周转数”(简称:k cat )。在科学杂志《自然通讯》上,杜塞尔多夫海因里希·海涅大学 (HHU) 的生物信息学家团队现在描述了一种使用人工智能方法预测各种酶的该参数的工具。
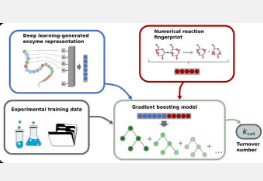
酶是所有活细胞中重要的生物催化剂。它们通常是大蛋白质,结合较小的分子(所谓的底物),然后将它们转化为其他分子,即“产物”。如果没有酶,将底物转化为产物的反应就无法发生,或者只能以非常低的速率进行。大多数生物体拥有数千种不同的酶。酶在各种生物技术过程和日常生活中有着广泛的应用——从面包面团的发酵到洗涤剂。
特定酶将其底物转化为产物的最大速度由所谓的周转数k cat决定。它是酶活性定量研究的重要参数,在理解细胞代谢方面发挥着关键作用。
然而,在实验中确定k只猫的周转数既耗时又昂贵,这就是为什么绝大多数反应都不为人所知。由 Martin Lercher 教授领导的 HHU 计算细胞生物学研究小组现在开发了一种名为 TurNuP 的新工具,可以使用人工智能方法预测酶的k cat周转数。
为了训练k cat预测模型,使用深度学习模型将有关酶和催化反应的信息转换为数值向量。这些数值向量作为机器学习模型(即所谓的梯度增强模型)的输入,该模型可预测 k只猫的周转次数。
主要作者 Alexander Kroll:“TurNuP 优于以前的模型,甚至可以成功用于与训练数据集中相似度较低的酶。” 除非至少 40% 的酶序列与训练集中的至少一种酶相同,否则之前的模型无法做出任何有意义的预测。相比之下,TurNuP 已经可以对最大序列同一性为 0 – 40% 的酶做出有意义的预测。
Lercher 教授补充道:“在我们的研究中,我们表明 TurNuP 所做的预测可用于比迄今为止的情况更准确地预测活细胞中的酶浓度。”
为了使尽可能多的用户可以轻松访问预测模型,HHU 团队开发了一个用户友好的网络服务器,其他研究人员可以使用它来预测酶的 k cat周转次数。
背景:机器学习和深度学习
深度学习模型由多层人工神经网络组成,可以识别和处理输入数据中的模式。使用大型训练数据集是训练深度学习模型处理数值输入的最佳方法。
梯度提升模型是一种机器学习方法,它会产生大量决策树。特定输入的所有决策树的结果用于进行预测。与深度学习类似,训练数据用于细化模型,即生成决策树。
免责声明:本文由用户上传,与本网站立场无关。财经信息仅供读者参考,并不构成投资建议。投资者据此操作,风险自担。 如有侵权请联系删除!
-
2025年6月20日,——在世界文化遗产地河南洛阳的光影流转之间,2025年新浪微博旅游之夜盛大举行。作为国内首个...浏览全文>>
-
2025年6月20日,——在世界文化遗产地河南洛阳的光影流转之间,2025年新浪微博旅游之夜盛大举行。作为国内首个...浏览全文>>
-
QQ多米试驾线下预约活动为了让更多用户感受QQ多米的独特魅力,我们特别推出了线下试驾预约活动。这不仅是一次...浏览全文>>
-
阜阳长安启源A07以其卓越的性能和豪华配置吸引了众多消费者的目光。作为一款定位高端市场的新能源车型,长安启...浏览全文>>
-
【安徽淮南大众CC新车报价2025款大公开】大众CC作为一款兼具运动感与豪华质感的轿跑车型,一直深受消费者喜爱...浏览全文>>
-
2025款长安猎手K50在安徽淮南地区的最新价格已新鲜出炉,为准备购车的朋友带来全面解析。这款车型以其高性价比...浏览全文>>
-
在安徽滁州购买长安猎手K50时,了解其落地价和省钱技巧至关重要。长安猎手K50是一款实用性强的皮卡车型,适合...浏览全文>>
-
途锐新能源是大众旗下的一款高端插电混动SUV,目前在安徽阜阳地区有售。其官方指导价约为58万元起,但实际成交...浏览全文>>
-
2025款大众CC作为一款兼具运动与豪华的中型轿车,备受关注。目前市场指导价大约在25万至35万元之间,具体价格...浏览全文>>
-
2024款探岳X作为一款备受关注的中型SUV,在市场上以其时尚的设计和出色的性能吸引了众多消费者。根据最新市场...浏览全文>>
- QQ多米试驾线下预约
- 安徽滁州长安猎手K50落地价,买车省钱秘籍
- 淮南大众CC新款价格2025款多少钱?买车攻略一网打尽
- 瑞虎8 PRO试驾,畅享豪华驾乘,体验卓越性能
- 安徽阜阳长安启源A05多少钱 2025款落地价,换代前的购车良机,不容错过
- 保时捷Macan试驾的流程是什么
- 安徽淮南大众ID.3多少钱?购车攻略在此
- 阜阳揽巡落地价,豪华配置超值价来袭
- 安徽池州威然 2024新款价格与配置的完美平衡
- 奇瑞瑞虎9试驾,新手必知的详细步骤
- QQ多米价格,换代前的购车良机,不容错过
- 池州迈腾GTE新款价格2022款多少钱?选车秘籍与优惠全公开
- 岚图追光多少钱 2024款落地价走势,近一个月最低售价25.28万起,性价比凸显
- 天津滨海威然 2024新款价格,最低售价28.98万起,入手正当时
- 蚌埠途昂新款价格2025款多少钱?购车必看
- 坦克400预约试驾全攻略
- 天津滨海ID.7 VIZZION价格,各配置车型售价全揭晓,性价比之王
- 安庆帕萨特最新价格2025款,最低售价12.35万起,入手正当时
- 亳州宝来新款价格2025款多少钱?选车指南与落地价全解析
- 生活家PHEV 2025新款价格,最低售价63.98万起现在该入手吗?
